
The French version of this document is no longer maintained: be sure to check the more up-to-date English version.
Graphiques
LaTeX
GUI
Dans cette partie (qui se veut plutôt une référence, et que l'on peut donc omettre en première lecture), nous nous intéressons à tous les détails que l'on peut configurer dans un graphique : choix des symboles, des couleurs, annotations (texte et symboles mathématiques), mais nous évoquerons aussi l'interfaçage avec LaTeX et la réalisation d'interfaces utilisateur graphiques avec Tk.
A FAIRE : c'est aussi ici qu'il faut dire comment produire des images PNG ou PDF.
Voici les principales commandes permettant de représenter des données. Nous ne les détaillons pas ici, nous le ferons plus loin.
La commande plot permet de tracer un ensemble de points, éventuellement en les reliant par une ligne.
data(beav1) plot(beav1$temp ~ beav1$time)

C'est en fait une fonction générique : elle se comportera différemment selon le type de son argument. Si son argument a pour classe "toto", R cherchera une fonction plot.toto ; si elle n'existe pas, il se rabattra sur la fonction plot.default. Les commandes plot, summary, predict (entre autres) fonctionnent sur ce principe.
> plot
function (x, y, ...)
{
if (is.null(attr(x, "class")) && is.function(x)) {
if ("ylab" %in% names(list(...)))
plot.function(x, ...)
else plot.function(x, ylab = paste(deparse(substitute(x)),
"(x)"), ...)
}
else UseMethod("plot")
}
> apropos("^plot\.")
[1] "plot.data.frame" "plot.default" "plot.density" "plot.factor"
[5] "plot.formula" "plot.function" "plot.histogram" "plot.lm"
[9] "plot.mlm" "plot.mts" "plot.new" "plot.POSIXct"
[13] "plot.POSIXlt" "plot.table" "plot.ts" "plot.TukeyHSD"
[17] "plot.window" "plot.xy"
> methods(plot)
[1] "plot.correspondence" "plot.lda" "plot.mca"
[4] "plot.profile" "plot.ridgelm" "plot.data.frame"
[7] "plot.default" "plot.density" "plot.factor"
[10] "plot.formula" "plot.function" "plot.histogram"
[13] "plot.lm" "plot.mlm" "plot.mts"
[16] "plot.new" "plot.POSIXct" "plot.POSIXlt"
[19] "plot.table" "plot.ts" "plot.TukeyHSD"
[22] "plot.window" "plot.xy"La commande matplot permet de tracer plusieurs courbes en même temps.
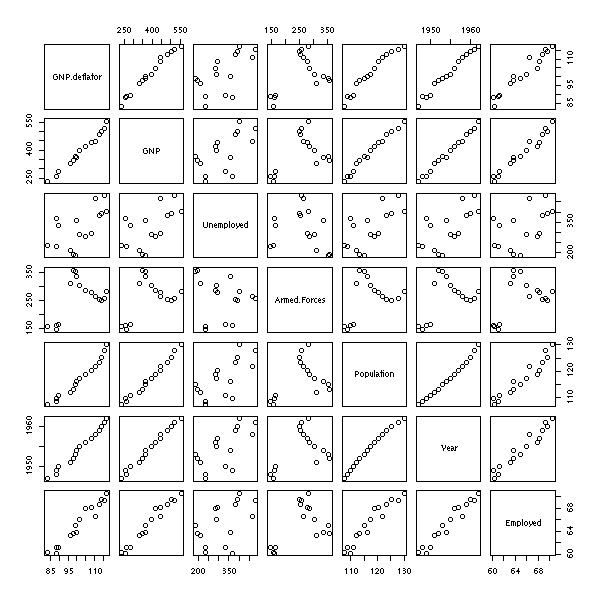
La commande pairs permet de tracer un tableau de nuages de points.
data(longley) pairs(longley)
La commande stripchart permet de tracer un << nuage >> de points unidimensionnel.
stripchart(longley$Unemployed)

La commande hist permet de tracer un histogramme.
hist(longley$Unemployed)

La commande boxplot permet de tracer un (ou plusieurs) diagrammes à moustaches.
boxplot(longley$Unemployed)

La commande contour permet de tracer des lignes de niveau.
La commande persp permet de tracer une surface.
Voici quelques manières de configurer ces graphiques.
On peut ajouter un titre, modifier le nom des axes.
n <- 100 x <- rnorm(n) y <- 1 - x^2 + .3*rnorm(n) plot(y~x, xlab='abscisses', ylab="ordonnées", main="Titre")

Dans ces titres, on peut mettre des symboles mathématiques.
x <- seq(-5,5,length=200)
y <- sqrt(1+x^2)
plot(y~x, type='l',
ylab=expression( sqrt(1+x^2) ))
title(main=expression("graphe de la fonction f"(x) == sqrt(1+x^2)))
On peut aussi prendre une telle formule et en changer un morceau (typiquement, pour y mettre la valeur d'une variable).
x <- seq(-5,5,length=200)
#plot(0,0, xlim=c(-5,5), ylim=c(0,6), type='n',xlab='', ylab='')
op <- par(mfrow=c(2,2))
for (i in 1:4) {
y <- sqrt(i+x^2)
plot(y~x, type='l', ylim=c(0,6),
ylab=substitute( expression( sqrt(i+x^2) ), list(i=i) ))
title(main=substitute("graphe de la fonction f"(x) == sqrt(i+x^2),
list(i=i)))
}
par(op)
Voici quelques exemples du manuel (on remarquera que le symbole sigma disparait...).
plot(1:10, 1:10, main = "text(...) examples\n~~~~~~~~~~~~~~",
sub = "R is GNU ©, but not ® ...")
mtext("«ISO-accents»: ± éè øØ å<Å æ<Æ", side=3)
points(c(6,2), c(2,1), pch = 3, cex = 4, col = "red")
text(6, 2, "the text is CENTERED around (x,y) = (6,2) by default",
cex = .8)
text(2, 1, "or Left/Bottom - JUSTIFIED at (2,1) by `adj = c(0,0)'",
adj = c(0,0))
text(4, 9, expression(hat(beta) == (X^t * X)^{-1} * X^t * y))
text(4, 8.4, "expression(hat(beta) == (X^t * X)^{-1} * X^t * y)", cex = .75)
text(4, 7, expression(bar(x) == sum(frac(x[i], n), i==1, n)))
Si on veut, on peut même écrire en japonais.
?Japanese
# Exemple du manuel
plot(1:9, type="n", axes=FALSE, frame=TRUE, ylab="",
main= "example(Japanese)", xlab= "using Hershey fonts")
par(cex=3)
Vf <- c("serif", "plain")
text(4, 2, "\\#J2438\\#J2421\\#J2451\\#J2473", vfont = Vf)
text(4, 4, "\\#J2538\\#J2521\\#J2551\\#J2573", vfont = Vf)
text(4, 6, "\\#J467c\\#J4b5c", vfont = Vf)
text(4, 8, "Japan", vfont = Vf)
par(cex=1)
text(8, 2, "Hiragana")
text(8, 4, "Katakana")
text(8, 6, "Kanji")
text(8, 8, "English")
# Autre exemple du manuel (qui contient aussi des tables
# de katakana et de kanji)
make.table <- function(nr, nc) {
opar <- par(mar=rep(0, 4), pty="s")
plot(c(0, nc*(10%/%nc) + 1), c(0, -(nr + 1)),
type="n", xlab="", ylab="", axes=FALSE)
invisible(opar)
}
get.r <- function(i, nr) i %% nr + 1
get.c <- function(i, nr) i %/% nr + 1
Esc2 <- function(str) paste("\\", str, sep="")
draw.title <- function(title, nc)
text((nc*(10%/%nc) + 1)/2, 0, title, font=2)
draw.vf.cell <- function(typeface, fontindex, string, i, nr, raw.string=NULL) {
r <- get.r(i, nr)
c <- get.c(i, nr)
x0 <- 2*(c - 1)
if (is.null(raw.string)) raw.string <- Esc2(string)
text(x0 + 1.1, -r, raw.string, col="grey")
text(x0 + 2, -r, string, vfont=c(typeface, fontindex))
rect(x0 + .5, -(r - .5), x0 + 2.5, -(r + .5), border="grey")
}
draw.vf.cell2 <- function(string, alt, i, nr) {
r <- get.r(i, nr)
c <- get.c(i, nr)
x0 <- 3*(c - 1)
text(x0 + 1.1, -r, Esc2(string <- Esc2(string)), col="grey")
text(x0 + 2.2, -r, Esc2(Esc2(alt)), col="grey", cex=.6)
text(x0 + 3, -r, string, vfont=c("serif", "plain"))
rect(x0 + .5, -(r - .5), x0 + 3.5, -(r + .5), border="grey")
}
tf <- "serif"
fi <- "plain"
nr <- 25
nc <- 4
oldpar <- make.table(nr, nc)
index <- 0
digits <- c(0:9,"a","b","c","d","e","f")
draw.title("Hiragana : \\\\#J24nn", nc)
for (i in 2:7) {
for (j in 1:16) {
if (!((i == 2 && j == 1) || (i == 7 && j > 4))) {
draw.vf.cell(tf, fi, paste("\\#J24", i, digits[j], sep=""),
index, nr)
index <- index + 1
}
}
}
On peut choisir la couleur des traits,
plot(runif(5), ylim=c(0,1), type='l')
for (i in c('red', 'blue', 'green')) {
lines( runif(5), col=i )
}
title(main="différentes couleurs de trait")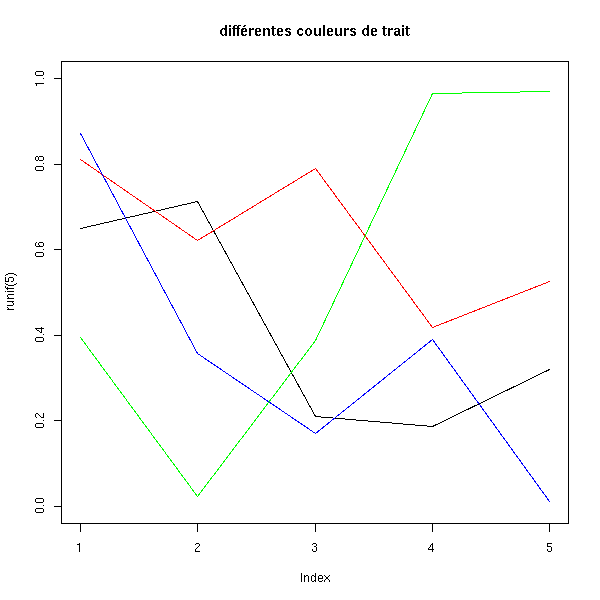
leur épaisseur,
plot(runif(5), ylim=c(0,1), type='n')
for (i in 5:1) {
lines( runif(5), col=i, lwd=i )
}
title(main="différentes épaisseurs de trait")
le type de tracé,
op <- par(mfrow=c(3,2)) plot(runif(5), type='p', main="plot type 'p' (points)") plot(runif(5), type='l', main="plot type 'l' (lines)") plot(runif(5), type='b', main="plot type 'b' (both points and lines)") plot(runif(5), type='s', main="plot type 's' (stair steps)") plot(runif(5), type='h', main="plot type 'h' (histogram)") plot(runif(5), type='n', main="plot type 'n' (no plot)") par(op)

les symboles utilisés (pch) ou leur taille (cex).
plot(0,0, xlim=c(1,5), ylim=c(-.5,4),
axes=F, xlab='', ylab='',
main="Symboles disponibles")
for (i in 0:4) {
for (j in 1:5) {
n <- 5*i+j
points(j,i, pch=n, cex=3)
text(j,i-.25, as.character(n))
}
}
Les options density et angle permettent de hachurer certaines zônes du dessin.
hist(longley$Unemployed, density=3, angle=45)

On peut mettre plusieurs graphiques dans un seul dessin. Il est alors parfois nécessaire de modifier les marges pour pouvoir ajouter un titre.
op <- par(mfrow = c(2, 2))
for (i in 1:4)
plot(runif(20), runif(20),
main=paste("random plot (",i,")",sep=''))
par(op)
mtext("Four plots, without enough room for this title",
side=3, font=2, cex=2, col='red')
op <- par(mfrow = c(2, 2), oma=c(0,0,3,0))
for (i in 1:4)
plot(runif(20), runif(20),
main=paste("random plot (",i,")",sep=''))
par(op)
mtext("Four plots, with some room for this title",
side=3, line=1.5, font=2, cex=2, col='red')
On peut même superposer des graphiques n'importe où...
n <- 20
x <- rnorm(n)
y <- x^2 - 1 + .3*rnorm(n)
plot(y~x)
op <- par()
for (i in 2:10) {
done <- FALSE
while(!done) {
a <- c( sort(runif(2,0,1)),
sort(runif(2,0,1)) )
par(fig=a, new=T)
r <- try(plot(runif(5), type='l', col=i))
done <- !inherits(r, "try-error")
}
}
par(op)
On peut superposer plusieurs éléments graphiques : soit à l'aide de commandes particulières (points, lines, segments, text), soit en ajoutant l'argument add=T à certaines de ces commandes, soit à l'aide de la commande par(new=T) (qui demande justement de ne pas commencer un nouveau dessin).
plot(runif(5), runif(5), xlim=c(0,1), ylim=c(0,1)) points(runif(5), runif(5), col='orange', pch=16, cex=3) lines(runif(5), runif(5), col='red') segments(runif(5), runif(5), runif(5), runif(5), col='blue') title(main="Superpositions...")

On peut colorier des parties du dessin à l'aide de la commande polygon.
my.col <- function (f, g, xmin, xmax, col, N=200) {
x <- seq(xmin,xmax,length=N)
fx <- f(x)
gx <- g(x)
plot(0,0, type='n',
xlim=c(xmin,xmax),
ylim=c( min(fx,gx), max(fx,gx) ) )
polygon( c(x,rev(x)), c(fx,rev(gx)), col='red', border=0 )
lines(x,fx,lwd=3)
lines(x,gx,lwd=3)
}
my.col( function(x) x^2, function(x) x^2+10*sin(x), -6, 6)
On peut définir des couleurs à l'aide d'un numéro, à l'aide d'une chaine de caractères contenant le nom de la couleur ou à l'aide d'une chaine de caractères contenant son codage RGB (comme "#CC00FF").
Les commandes rainbow, heat.color, etc. permettent d'obtenir facilement des listes de couleurs.
# Exemple du manuel
ch.col <- c("rainbow(n, start=.7, end=.1)", "heat.colors(n)",
"terrain.colors(n)", "topo.colors(n)", "cm.colors(n)")
n <- if(.Device == "postscript") 64 else 16
# Since for screen, larger n may give color allocation problem
nt <- length(ch.col)
i <- 1:n; j <- n / nt; d <- j/6; dy <- 2*d
plot(i,i+d, type="n", yaxt="n", ylab="", main=paste("color palettes; n=",n))
for (k in 1:nt) {
rect(i-.5,(k-1)*j+ dy, i+.4, k*j, col=eval(parse(text=ch.col[k])))
text(2*j, k * j +dy/4, ch.col[k])
}
La commande legend permet d'ajouter une légende au dessin.
x <- seq(-6,6,length=200)
y <- sin(x)
z <- cos(x)
plot(y~x, type='l', lwd=3,
ylab='', xlab='angle', main="Fonctions trigonométriques")
abline(h=0,lty=3)
abline(v=0,lty=3)
lines(z~x, type='l', lwd=3, col='red')
legend(-6,-1, yjust=0,
c("Sinus", "Cosinus"),
lwd=3, lty=1, col=c(par('fg'), 'red'),
)
Pour la positionner précisément, on peut utiliser les limites du dessin.
xmin <- par('usr')[1]
xmax <- par('usr')[2]
ymin <- par('usr')[3]
ymax <- par('usr')[4]On peut aussi la positionner interactivement, à l'aide de la commande locator, qui demande à l'utilisateur de cliquer sur une partie du dessin et qui renvoie les coordonnées du point ainsi désigné.
A FAIRE : rédiger, donner un exemple.
labcurve in Hmisc : to put the name of the curve on the curve (and not in a remote legend)
On peut ajouter une grille derrière un dessin, pour faciliter sa lecture.
?grid
On peut aussi faire le dessin sur un fond (un dégradé ou une image éclaircie) : il suffit de sauvegarder le graphique au format PNG, avec un fond transparent, et le mettre sur l'image voulue à l'aide de la commande composite (dans ImageMagick).
composite faithful_hist_orig.png faithful600_light.jpg faithful_hist.png

La commande locator permet d'obtenir les coordonnées d'un point en cliquant dessus, la commande identify permet d'avoir le nom d'un point en cliquant dessus.
Pour plus d'interactivité, on peut utiliser Tk ou programme exterme (ainsi nous parlerons plus loin de xgobi).
Une bibliothèque de bas niveau pour faire des graphiques, qui remplace complètement les commandes que nous venons de voir. C'est ce qui est utilisé, de manière interne, par la bibliothèque lattice.
library(help=grid) library(grid) ?Grid
L'idée est de diviser la zône de dessin en parties rectangulaires (qui peuvent éventuellement se superposer) : les Viewports.
Les commandes pour tracer des segments, des points ne sont plus les mêmes, mais s'appellent désormais : grid.lines, grid.points, etc.
> apropos("^grid\.")
[1] "grid.Call" "grid.Call.graphics" "grid.circle"
[4] "grid.collection" "grid.copy" "grid.display.list"
[7] "grid.draw" "grid.edit" "grid.frame"
[10] "grid.get" "grid.grill" "grid.grob"
[13] "grid.layout" "grid.legend" "grid.lines"
[16] "grid.line.to" "grid.move.to" "grid.multipanel"
[19] "grid.newpage" "grid.pack" "grid.panel"
[22] "grid.place" "grid.plot.and.legend" "grid.points"
[25] "grid.polygon" "grid.pretty" "grid.prop.list"
[28] "grid.rect" "grid.segments" "grid.set"
[31] "grid.show.layout" "grid.show.viewport" "grid.strip"
[34] "grid.text" "grid.top.level.vp" "grid.xaxis"
[37] "grid.yaxis"On n'obtient plus les paramétres graphiques par la commande par(), mais avec gpar(). Quand on veut modifier les valeurs de certains de ces paramètres, on ajoute un argument gpar à la commande en question.
grid.rect(gp=gpar(fill="grey"))
Les lignes suivantes commencent un nouveau dessin, le colorient en gris, créent un nouveau viewport et de placent dedans.
grid.newpage() grid.rect(gp=gpar(fill="grey")) push.viewport(...) ... pop.viewport()
On peut définir un Viewport par sa largeur et sa hauteur (par rapport au viewport qui le contient). On peut aussi le munir d'unités (quand on fera des dessins dans ce Viewport, on pourra utiliser soit les unités par défaut (largeur=hauteur=1), soit ces unités-utilisateur, à l'aide de la commande unit).
viewport(w = 0.9, h = 0.9, # largeur et hauteur
xscale=c(xmin,xmax),
yscale=c(ymin,ymax),
)Généralement, on ajoute un peu de blanc à l'intérieur du cadre :
viewport(w = 0.9, h = 0.9,
xscale=c(xmin,xmax)+.05*c(-1,1),
yscale=c(ymin,ymax)+.05*c(-1,1),
)Voici un exemple du manuel.
library(grid)
grid.show.viewport(viewport(x=0.6, y=0.6,
w=unit(1, "inches"), h=unit(1, "inches")))
On peut aussi définir un Viewport contenant un tableau de Viewports.
viewport(layout=grid.layout(2, 2)))
Voici un exemple du manuel. La commande suivante définit 8 Viewports, qui sont représentés par les cases bleues sur le dessin (la partie en blanc restera blanche).
grid.show.layout(grid.layout(4,2,
heights=unit(rep(1, 4),
c("lines", "lines", "lines", "null")),
widths=unit(c(1, 1), "inches")))
Les lignes suivantes commencent un nouveau dessin, définissent un nouveau viewport, le divisent en quatre et mettent quelque chose dans chacun des quatre morceaux.
dessine <- function () {
push.viewport(viewport(w = 0.9, h = 0.9,
xscale=c(-.1,1.1), yscale=c(-.1,1.1)))
grid.rect(gp=gpar(fill=rgb(.5,.5,0)))
grid.points( runif(50), runif(50) )
pop.viewport()
}
grid.newpage()
grid.rect(gp=gpar(fill=rgb(.3,.3,.3)))
push.viewport(viewport(layout=grid.layout(2, 2)))
for (i in 1:2) {
for (j in 1:2) {
push.viewport(viewport(layout.pos.col=i,
layout.pos.row=j))
dessine()
pop.viewport()
}
}
pop.viewport()
Parmi les exemples du manuel, on trouve :
grid.multipanel(vp=viewport(0.5, 0.5, 0.8, 0.8))

Mais cette fonction n'est pas documentée. Néanmoins, son code est assez instructif.
> grid.multipanel
function (x = runif(90), y = runif(90), z = runif(90), nrow = 2,
ncol = 5, nplots = 9, newpage = TRUE, vp = NULL)
{
# Si c'est demandé, on commence un nouveau dessin
if (newpage)
grid.newpage()
# Si on a précisé, en argument, qu'il fallait se placer
# dans un certain Viewport, on le fait -- sinon, on reste
# dans le Viewport courrant, i.e., toute la page.
# (Dans l'exemple précédent, on avait mis un argument, de
# manière à avoir une marge autour du dessin.)
if (!is.null(vp))
push.viewport(vp)
# On crée un Viewport, qu'on divise en un tableau de Viewports.
# On constate ici que la fonction grid.multipanel a été écrite
# pour servir d'illustration, car c'est l'utilisateur qui doit
# fixer le nombre de lignes et de colonnes : on lui fait confiance
# pour que les dessins aient un format lisible et pour que le
# tableau ait suffisemment de cases...
temp.vp <- viewport(layout = grid.layout(nrow, ncol))
push.viewport(temp.vp)
# On calcule les unités-utilisateur :
# les limites de x et y, plus 2.5% de chaque côté
xscale <- range(x) + c(-1, 1) * 0.05 * diff(range(x))
yscale <- range(y) + c(-1, 1) * 0.05 * diff(range(y))
breaks <- seq(min(z), max(z), length = nplots + 1)
for (i in 1:nplots) {
col <- (i - 1)%%ncol + 1
row <- (i - 1)%/%ncol + 1
# On se place ensuite dans chacune des cases du tableau
panel.vp <- viewport(layout.pos.row = row, layout.pos.col = col)
# Dans chacune des cases, on ne trace qu'un petit morceau
# des données : on regarde ici quels sont les points concernés.
panelx <- x[z >= breaks[i] & z <= breaks[i + 1]]
panely <- y[z >= breaks[i] & z <= breaks[i + 1]]
# On donne tout ça à la fonction grid.panel, qui fait le travail...
grid.panel(panelx, panely, range(z),
c(breaks[i], breaks[i + 1]),
xscale, yscale,
axis.left = (col == 1),
axis.left.label = is.odd(row),
axis.right = (col == ncol || i == nplots),
axis.right.label = is.even(row),
axis.bottom = (row == nrow),
axis.bottom.label = is.odd(col),
axis.top = (row == 1),
axis.top.label = is.even(col),
vp = panel.vp)
}
# On utilise la commande grid.text pour nommer les axes
grid.text("Compression Ratio",
unit(0.5, "npc"), unit(-4, "lines"),
gp = gpar(fontsize = 20),
just = "center", rot = 0)
grid.text("NOx (micrograms/J)",
unit(-4, "lines"), unit(0.5, "npc"),
gp = gpar(fontsize = 20),
just = "centre", rot = 90)
pop.viewport()
if (!is.null(vp))
pop.viewport()
}Regardons maintenant le code de la fonction grid.panel :
> grid.panel
function (x = runif(10), y = runif(10),
zrange = c(0, 1), zbin = runif(2),
xscale = range(x) + c(-1, 1) * 0.05 * diff(range(x)),
yscale = range(y) + c(-1, 1) * 0.05 * diff(range(y)),
axis.left = TRUE, axis.left.label = TRUE,
axis.right = FALSE, axis.right.label = TRUE,
axis.bottom = TRUE, axis.bottom.label = TRUE,
axis.top = FALSE, axis.top.label = TRUE,
vp = NULL)
{
if (!is.null(vp))
push.viewport(vp)
# On découpe le Viewport : une ligne en haut (pour le nom de la classe)
# et le reste en dessous.
temp.vp <- viewport(layout =
grid.layout(2, 1, heights = unit(c(1, 1), c("lines", "null"))))
push.viewport(temp.vp)
strip.vp <- viewport(layout.pos.row = 1, layout.pos.col = 1,
xscale = xscale)
push.viewport(strip.vp)
# La commande grid.strip dessine un rectangle orange clair,
# représentant toute l'étendue des données, auquel se superpose
# un rectangle orange fonçé, représentant l'étendue des données
# représentées dans ce Viewport
grid.strip(range.full = zrange, range.thumb = zbin)
# Un cadre noir autour
grid.rect()
if (axis.top)
grid.xaxis(main = FALSE, label = axis.top.label)
pop.viewport()
# On passe au dessin lui-même
plot.vp <- viewport(layout.pos.row = 2, layout.pos.col = 1,
xscale = xscale, yscale = yscale)
push.viewport(plot.vp)
# On dessine une grille
grid.grill()
# On trace les points
grid.points(x, y, gp = gpar(col = "blue"))
# Un cadre autour du dessin
grid.rect()
# Les axes, si c'est demandé
if (axis.left)
grid.yaxis(label = axis.left.label)
if (axis.right)
grid.yaxis(main = FALSE, label = axis.right.label)
if (axis.bottom)
grid.xaxis(label = axis.bottom.label)
pop.viewport(2)
if (!is.null(vp))
pop.viewport()
invisible(list(strip.vp = strip.vp, plot.vp = plot.vp))
}Voici un autre exemple :
do.it <- function (x=runif(100), y=runif(100),
a=.9, b=.1,
col1=rgb(0,.3,0), col2=rgb(1,1,0)) {
xscale <- range(x) + c(-1,1)*.05
yscale <- range(y) + c(-1,1)*.05
grid.newpage()
grid.rect(gp=gpar(fill=col1, col=col1))
w1 <- a - b/2
w2 <- 1 - a - b/2
c1 <- b/3 + w1/2
c2 <- a + b/6 + w2/2
vp1 <- viewport(x=c1, y=c1, width=w1, height=w1,
xscale=xscale, yscale=yscale)
push.viewport(vp1)
grid.rect(gp=gpar(fill=col2, col=col2))
grid.points(x,y)
pop.viewport()
vp2 <- viewport(x=c1, y=c2, width=w1, height=w2,
xscale=xscale, yscale=c(0,1))
push.viewport(vp2)
grid.rect(gp=gpar(fill=col2, col=col2))
grid.points(x,rep(.5,length(x)))
pop.viewport()
vp3 <- viewport(x=c2, y=c1, width=w2, height=w1,
xscale=c(0,1), yscale=yscale)
push.viewport(vp3)
grid.rect(gp=gpar(fill=col2, col=col2))
grid.points(rep(.5,length(y)),y)
pop.viewport()
}
do.it()
Exercice : Remplacer les diagrammes de dispersion par des boites à moustaches.
La bibliothèque xtable permet de transformer les tables produites par R (en particulier les tables d'analyse de variance) en tableaux LaTeX, que l'on peut ensuite inclure dans un article.
> xtable(anova(lm.D9 <- lm(weight ~ group)))
% latex table generated in R 1.6.2 by xtable 1.0-11 package
% Fri Feb 28 18:47:58 2003
\begin{table}
\begin{center}
\begin{tabular}{lrrrrr}
\hline
& Df & Sum Sq & Mean Sq & F value & Pr($>$F) \\
\hline
group & 1 & 0.69 & 0.69 & 1.42 & 0.2490 \\
Residuals & 18 & 8.73 & 0.48 & & \\
\hline
\end{tabular}
\end{center}
\end{table}
> xtable(summary(lm(weight~group)))
% latex table generated in R 1.6.2 by xtable 1.0-11 package
% Fri Feb 28 18:57:00 2003
\begin{table}
\begin{center}
\begin{tabular}{rrrrr}
\hline
& Estimate & Std. Error & t value & Pr($>$$|$t$|$) \\
\hline
(Intercept) & 5.0320 & 0.2202 & 22.85 & 0.0000 \\
groupTrt & $-$0.3710 & 0.3114 & $-$1.19 & 0.2490 \\
\hline
\end{tabular}
\end{center}
\end{table}
Voir aussi la commande latex dans la bibliothèque Hmisc.
Sweave permet de mettre du code R dans un document LaTeX, de sorte qu'à chaque compilation, R refasse les calculs et les dessins. C'est utile si on rédige une documentation de R (de la sorte, on est sûr que le code qu'on donne est correct et qu'il correspond aux dessins) ou de manière plus générale si on mentionne les résultats de calculs avec R dans l'article (par exemple, une table d'Anova) ou si on l'illustre de graphiques produits par R.
library(tools) ?Sweave http://www.ci.tuwien.ac.at/~leisch/Sweave
Voici un exemple :
\documentclass[a4paper,12pt]{article}
\usepackage[T1]{fontenc}
\usepackage[latin1]{inputenc}
\usepackage[frenchb]{babel}
\parindent=0pt
\begin{document}
Des calculs dont on n'affiche ni le code, ni le résultat :
<<results=hide,echo=FALSE>>=
y <- 1+1
y
@
(il n'y a rien qui s'affiche, c'est normal).
Des calculs dont on n'affiche pas le résultat :
<<results=hide,echo=TRUE>>=
library("MASS")
ajoute <- function (a,b) {
a+b
}
x <- ajoute(1,2)
x
@
Des calculs dont on affiche le résultat :
<<results=verbatim>>=
n <- 100
x <- runif(n)
y <- 1 - x - x^2 + rnorm(n)
r <- lm(y~poly(x,5))
summary(r)
@
Des calculs dont le résultat est préalablement converti en TeX par R :
<<results=tex>>=
cat("\\centerline{\\LaTeX}")
@
Un graphique :
<<fig=TRUE>>=
n <- 100
a <- rnorm(n)
b <- 1 - 2*a + rnorm(n)
plot(b~a)
abline(lm(b~a), col='red', lwd=3)
@
<<results=hide,echo=FALSE>>=
a <- function (x) {
round(x, digits=2)
}
@
Il est aussi possible d'utiliser le résultat de certains calculs dans
le corps du texte : les coefficients de la première régression sont
\Sexpr{a(r$coef[1])}, \Sexpr{a(r$coef[2])} et \Sexpr{a(r$coef[3])}.
\end{document}On le compile ainsi :
Sweave("tmp.Rnw")
system("pdflatex tmp.tex")
system("xpdf tmp.pdf")Voivi le résultat :
SweaveExample.pdf
Les options qui apparaissent dans ce code sont détaillées dans :
?RweaveLatex
Il est possible de construire des interfaces graphiques directement sous R à l'aide de Tcl/Tk.
library(tcltk) library(help=tcltk)
Pour la documentation des Widgets, il faut aller voir ailleurs...
http://www.math.jussieu.fr/~zoonek/UNIX/10_ptk/1.html
Voici un exemple : une mini-machine à calculer.
tkdestroy(wtop)
wtop <- tktoplevel()
w.titre <- tklabel(wtop, text="Additions")
w.un <- tkentry(wtop)
w.deux <- tkentry(wtop)
w.resultat <- tklabel(wtop, text=0)
tkpack(w.titre, w.un, w.deux, w.resultat)
on.key.press <- function () {
# Que c'est compliqué !
a <- tclvalue(tkget(w.un))
a <- eval(parse(text=a))
if(!is.numeric(a)) a <- 0
b <- eval(parse(text=b))
b <- tclvalue(tkget(w.deux))
if(!is.numeric(b)) b <- 0
tkconfigure(w.resultat, text=a+b)
}
tkbind(wtop, "<KeyPress>", on.key.press)
Voici un exemple du manuel.
tkdestroy(tt) tt <- tktoplevel() tkpack(txt.w <- tktext(tt)) tkinsert(txt.w, "0.0", "plot(1:10)") eval.txt <- function() eval(parse(text=tclvalue(tkget(txt.w, "0.0", "end")))) tkpack(but.w <- tkbutton(tt,text="Submit", command=eval.txt))
Le lecteur intéressé regardera tout particulièrement la bibliothèque tkrplot, qui permet d'inclure un graphique R normal dans un widget. L'exemple du manuel montre une courbe, qui dépend d'un paramètre, que l'on peut fixer en faisant varier un assenseur.

Une fonction qui prend en argument une fonction qui trace un graphique en fonction d'un paramètre et les limites de variation de ce paramètre.
library(tkrplot)
animate <- function (plot.function, limits) {
bb <- mean(limits)
tt <- tktoplevel()
img <-tkrplot(tt, function () { plot.function(bb) } )
f <- function (...) {
b <- as.numeric(tclvalue("bb"))
if (b != bb) {
bb <<- b
tkrreplot(img)
}
}
s <- tkscale(tt, command=f, from=limits[1], to=limits[2], variable="bb",
showvalue=TRUE, resolution=diff(range(limits))/100, orient="horiz")
tkpack(img,s)
}
animate( function (a) { hist(abs(rnorm(200))^a) }, c(.1,2) )Exemple : Trouver la transformation à faire subir à une variable pour qu'elle redevienne normale.
n <- 200
k <- runif(1, 0,2)
x <- (5+rnorm(n))^k
animate( function (a) { x <- x^(1/a); qqnorm(x); qqline(x,col='red') },
c(.01,2) )Exemple : Regarder l'effet des changement de la largeur des classes sur un histogramme.
n <- 200
x <- rnorm(n)
animate( function (a) {
a <- ceiling(a)
print(a)
hist(x, breaks=a, col='light blue', probability=T);
lines(density(x), col='red', lwd=3)
},
c(2,102) )Exemple : Illustrer le théorème de la limite centrale (dont nous parlerons un peu plus loin) à l'aide d'une animation interactive.
N <- 1000
n <- 102
m <- .5
s <- 1/sqrt(12)
x <- matrix(runif(n*N), nc=n)
animate( function (a) {
x <- (apply(x[,1:a],1,sum) - a*m)/(sqrt(a)*s)
hist(x, col='light blue', probability=T, main=paste("n =",a), ylim=c(0,.4),
xlim=c(-4,4))
lines(density(x), col='red', lwd=3)
curve(dnorm(x), col='blue', lwd=3, lty=3, add=T)
if( N>100 ) {
rug(sample(x,100))
} else {
rug(x)
}
},
c(2,102)
)
# Idem, avec une distribution bimodale
N <- 1000
n <- 101
m <- 0
s <- sqrt(10)
x <- rnorm(n*N, sample(c(-3,3),n*N,replace=T))
x <- matrix(x, nc=n)
animate( function (a) {
x <- (apply(x[,1:a],1,sum) - a*m)/(sqrt(a)*s)
hist(x, col='light blue', probability=T, main=paste("n =",a), ylim=c(0,.4),
xlim=c(-4,4))
lines(density(x), col='red', lwd=3)
curve(dnorm(x), col='blue', lwd=3, lty=3, add=T)
if( N>100 ) {
rug(sample(x,100))
} else {
rug(x)
}
},
c(1,101)
)
# Idem, avec une dictribution asymétrique
# Idem, avec une distribution bimodale
N <- 1000
n <- 102
m <- 1
s <- 1
x <- rexp(n*N)
x <- matrix(x, nc=n)
animate( function (a) {
x <- (apply(x[,1:a],1,sum) - a*m)/(sqrt(a)*s)
hist(x, col='light blue', probability=T, main=paste("n =",a), ylim=c(0,.4),
xlim=c(-4,4))
lines(density(x), col='red', lwd=3)
curve(dnorm(x), col='blue', lwd=3, lty=3, add=T)
if( N>100 ) {
rug(sample(x,100))
} else {
rug(x)
}
},
c(1,101)
)Exemple : Représenter la densité d'une distribution dépendant d'un paramètre.
A FAIRE
A FAIRE : d'autres exemples (terminer de les rédiger)
écrire une fonction générale qui permet de modifier un graphique en faisant varier un paramètre (ou plusieurs). Applications : courbe de densité d'une loi à deux paramètres qqnorm et transformation d'une variable histogramme et densité : faire varier bw & offset Idem avec plusieurs graphiques, qui varient simultanément densité + fonction de répartition + qqnorm + boxplot d'une loi dépendant d'un ou plusieurs paramètres, ou d'un échantillon subissant une transformation dépendant d'un paramètre.
Vincent Zoonekynd
<zoonek@math.jussieu.fr>
latest modification on Wed Oct 13 22:32:32 BST 2004