The French version of this document is no longer maintained: be sure to check the more up-to-date English version.
Bidouillages
Persistance
Bibliothèques (library) supplémentaires
Programmation Orientée Objets
Autres langages
Dans cette partie (qui peut être omise en première lecture), nous nous intéressons à des caractéristiques plus pointues de la programmation sous R : débugguage, profiling, espaces de nommage, objets, interface avec d'autres programmes, avec des bases de données, avec d'autres langages.
La commande debug permet d'exécuter une fonction pas à pas.
> debug(f)
> f(3)
debugging in: f(3)
debug: {
x^2 + x + 1
}
Browse[1]>
debug: x^2 + x + 1
Browse[1]>
exiting from: f(3)
[1] 13
> undebug(f)La commande traceback imprime l'état de la pile des appels de fonctions lors de la dernière erreur (les fonctions les plus profondes en premier).
?traceback
La commande dump.frames permet de récupérer l'équivalent d'un fichier core (l'état de l'interpréteur à un instant donné, typiquement juste après une erreur), que l'on peut ensuite examiner à l'aide de la commande debugger.
?dump.frames
Il y a une commande try, pour exécuter des fonctions qui plantent parfois.
try(...)
Nous serons amenés à l'utiliser un peu plus loin dans ce document, souvent sous la forme
x <- NA
try( x <- ... )
if( is.na(x) ) {
...
} else {
...
}
Les fonctions n'ont pas d'effet de bord, donc on ne devrait pas pouvoir modifier des variables globales à l'intérieur d'une fonction. Néanmoins, la syntaxe
x <<- 1+2+3+4+5
modifie la variable x là où elle se trouve ou, si elle n'est pas encore définie, la définit comme variable globale.
Autre manière de procéder :
set.global <- function (x, value) {
x <- deparse(substitute(x))
assign(x, value, pos=.GlobalEnv)
}Et ça marche :
> set.global(a,3) > a [1] 3 > set.global(a,1:10) > a [1] 1 2 3 4 5 6 7 8 9 10
On peut généraliser cette fonction pour écrire des méthodes qui modifient l'objet auquel elles sont attachées. Au lieu d'utiliser pos=.GlobalEnv, on peut mettre -2, ce qui correspondra (je crois) à une variable locale à l'endroit d'où la fonction est appelée.
On peut créer explicitement des espaces de nommage ("environements") et ensuite les utiliser comme des tables de hachage.
> x <- new.env(hash=T)
> assign('foo', 3, env=x)
> assign('bar', list('a'=3, 'b'=list('c'=1, d='foo')), env=x)
> assign('baz', data.frame(rnorm(10),rnorm(10)), env=x)
> x
<environment: 0x8bb97bc>
> ls(env=x)
[1] "bar" "baz" "foo"
> get('foo', env=x)
[1] 3
On peut demander à R d'effectuer certaines actions lorsqu'on le lance (par exemple, charger une certaine bibliothèque) ou lorsqu'on le quitte (par exemple, sauvegarder les données sous un certain format), en redéfinissant les fonction .First() et .Last().
> ?Startup
> .First
function() {
require("ctest", quietly = TRUE)
}
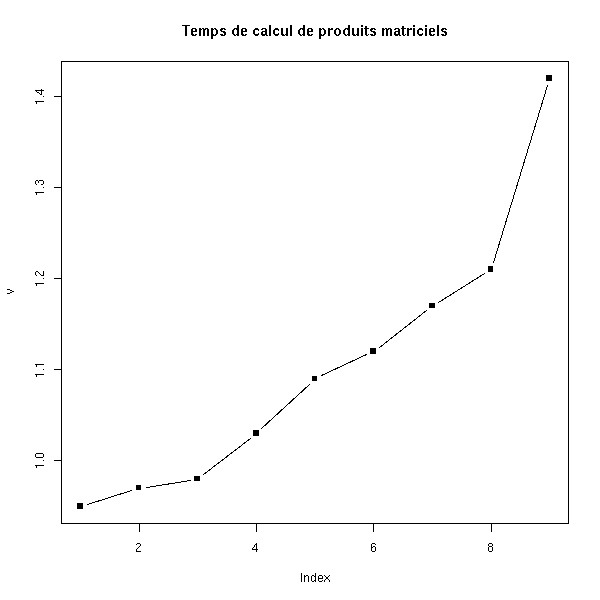
La commande system.time permet de savoir combien de temps prend une commande.
several.times <- function (n, f, ...) {
for (i in 1:n) {
f(...)
}
}
matrix.multiplication <- function (s) {
A <- matrix(1:(s*s), nr=s, nc=s)
B <- matrix(1:(s*s), nr=s, nc=s)
C <- A %*% B
}
v <- NULL
for (i in 2:10) {
v <- append(v,
system.time( several.times( 10000, matrix.multiplication, i )) [1])
}
plot(v, type='b', pch=15, main="Temps de calcul de produits matriciels")
La commande Rprof permet de voir dans quelle partie d'un programme ou d'une fonction on passe le plus de temps.
?Rprof
Exemple :
Rprof() n <- 200 m <- matrix(rnorm(n*n), nr=n, nc=n) eigen(m)$vectors[,c(1,2)] Rprof(NULL)
On examine ensuite le résultat (on n'est plus sous R, on appelle R avec d'autres options, depuis le shell) :
% R CMD Rprof Rprof.out Each sample represents 0.02 seconds. Total run time: 0.9 seconds. Total seconds: time spent in function and callees. Self seconds: time spent in function alone. % total % self total seconds self seconds name 95.56 0.86 2.22 0.02 "eigen" 82.22 0.74 82.22 0.74 ".Fortran" 11.11 0.10 4.44 0.04 "all.equal.numeric" 4.44 0.04 0.00 0.00 "matrix" 4.44 0.04 0.00 0.00 "as.vector" 4.44 0.04 4.44 0.04 "rnorm" 2.22 0.02 2.22 0.02 "<Anonymous>" 2.22 0.02 2.22 0.02 "|" 2.22 0.02 2.22 0.02 "t.default" 2.22 0.02 0.00 0.00 "mean" 2.22 0.02 0.00 0.00 "t" % self % total self seconds total seconds name 82.22 0.74 82.22 0.74 ".Fortran" 4.44 0.04 11.11 0.10 "all.equal.numeric" 4.44 0.04 4.44 0.04 "rnorm" 2.22 0.02 2.22 0.02 "<Anonymous>" 2.22 0.02 2.22 0.02 "|" 2.22 0.02 2.22 0.02 "t.default" 2.22 0.02 95.56 0.86 "eigen"
La commande system permet de lancer des commandes Unix quelconques.
system("top")Si on veut lancer la commande en tache de fond, on ajoute un & à la fin, comme d'habitude. Si la commande attend des arguments (en particulier un nom de fichier, contenant les données à traiter), il est probablement utile d'afficher exactement la commande sui sera utilisée. Ainsi, dans le code de xgobi (un programme extérieur, que l'on peut utiliser pour visualiser des nuages de points en dimension 3 ou plus), on trouve
...
args <- paste("-title", paste("'", title, "'", sep = ""),
args)
command <- paste("xgobi", args, dfile, "&")
cat(command, "\n")
s <- system(command, FALSE)
invisible(s)
}Cela sert principalement à passer les données sur lesquelles on travaille à un autre programme, souvent pour les visualiser. On verra ainsi comment appeler xgobi (un logiciel pour visualiser, de manière dynamique et interactive, des données multivariées) depuis R. On peut aussi l'utiliser pour produire de beaux dessins, par exemple ajouter une image en arrière plan d'un graphique PNG à l'aide d'ImageMagick, ou pour demander à PoVRay de faire un dessin (comme dans l'image qui sert de titre à ce document).
On peut aussi l'utiliser pour lancer automatiquement LaTeX (puis xdvi, dvips et même lpr) sur un fichier préalablement créé (avec SWeave, qui permet de mettre des calculs sous R et les éventuelles illustrations correspondantes dans un document LaTeX).
La commande deparse permet de voir le contenu d'un objet. Elle permet par exemple de transformer une fonction en une chaine de caractère pour l'imprimer dans un fichier LaTeX (si on veut juste l'imprimer à l'écran, il suffit de donner son nom à l'interpréteur, sans parenthèses).
> print
function (x, ...)
UseMethod("print")
> deparse(print)
[1] "function (x, ...) " "UseMethod(\"print\")"La commande substitute permet d'effectuer des remplacements dans une expression (le résultat est une expression non évaluée).
> substitute(x+1, list(x=3)) 3 + 1
Nous avons aussi vu que la commande substitute (éventuellement accompagnée de la commande deparse) permettait à une fonction de savoir d'où provenait l'un de ses arguments.
my.arg <- function (x, ...) {
cat("My first argument was: ")
cat(deparse(substitute(x)))
cat("\n")
}
> my.arg(3)
My first argument was: 3
> my.arg(x)
My first argument was: x
> my.arg(x+1)
My first argument was: x + 1La commande get permet de voir le contenu d'une variable dont le nom est contenu dans une chaine de caractères.
> get("plot")
function (x, ...)
{
if (is.null(attr(x, "class")) && is.function(x)) {
if ("ylab" %in% names(list(...)))
plot.function(x, ...)
else plot.function(x, ylab = paste(deparse(substitute(x)),
"(x)"), ...)
}
else UseMethod("plot")
}La commande ls permet d'obtenir la liste des variables définies. On peut préciser dans quel environement chercher (on peut ainsi chercher dans les variables globales, même si elles sont cachées par des variables locales). On peut aussi définir la recherche par une expression régulière.
> ls() [1] "a" "my.arg" "set.global" "x" "y" > ls(pos=.GlobalEnv) [1] "a" "my.arg" "set.global" "x" "y"
Comme sous Unix, les variables dont le nom commence par un point sont << cachées >> : il faut demander explicitement à les voir.
> ls(all=T) [1] "a" "my.arg" ".Random.seed" "set.global" [5] ".Traceback" "x" "y"
La commande rm permet d'effacer des variables.
La commande search permet de voir le chemin dans lequel R cherche les variables.
> library(MASS) > search() [1] ".GlobalEnv" "package:MASS" "package:ctest" "Autoloads" [5] "package:base" > ls(pos="package:MASS") [1] "addterm" "addterm.default" "addterm.glm" [4] "addterm.lm" "addterm.mlm" "addterm.negbin" [7] "addterm.survreg" "anova.loglm" "anova.negbin" ... [175] "update.loglm" "vcov" "vcov.glm" [178] "vcov.lm" "vcov.nls" "vcov.polr" [181] "width.SJ" "write.matrix"
La commande apropos (ou find) permet de trouver le nom d'une variable.
> apropos('exp')
[1] "negexp.SSival" "as.expression" "as.expression.default"
[4] "char.expand" "dexp" "exp"
[7] "expand.grid" "expand.model.frame" "expm1"
[10] "expression" "is.expression" "path.expand"
[13] "pexp" "qexp" "regexpr"
[16] "rexp"
> apropos('.')
[1] "x" "boxcox"
[3] "boxcox.default" "boxcox.formula"
[5] "boxcox.lm" "corresp.matrix"
...
[1767] "xyinch" "xyz.coords"
[1769] "yinch" "zapsmall"
[1771] "zip.file.extract"La commande stop permet d'arréter une fonction quand on s'apperçoit d'un problème (par exemple, à propos du type de ses arguments).
do.it <- function (x) {
if( !is.numeric(x) )
stop("Expecting a NUMERIC vector!")
if( !is.vector(x) )
stop("Expecting a numeric VECTOR!")
if( length(x)<2 )
stop("Expecting a numeric vector of length at least 2")
return("Well done.")
}
> do.it("abc")
Error in do.it("abc") : Expecting a NUMERIC vector!
> do.it(3)
Error in do.it(3) : Expecting a numeric vector of length at least 2
> do.it(data.frame(a=1:3,b=3:1))
Error in do.it(data.frame(a = 1:3, b = 3:1)) :
Expecting a NUMERIC vector!
> do.it(matrix(1:4,nc=2,nr=2))
Error in do.it(matrix(1:4, nc = 2, nr = 2)) :
Expecting a numeric VECTOR!
> do.it(1:26)
[1] "Well done."La commande parse permet de transformer une chaine de caractères en une expression. On peut ensuite l'évaluer à l'aide de la commande eval.
> parse(text="0==1") expression(0 == 1) > eval(parse(text="0==1")) [1] FALSE
Les expressions peuvent aussi s'utiliser comme labels, dans les graphiques.
x <- seq(0,4, length=100) y <- sqrt(x) plot(y~x, type='l', lwd=3, main=expression(y == sqrt(x)) )
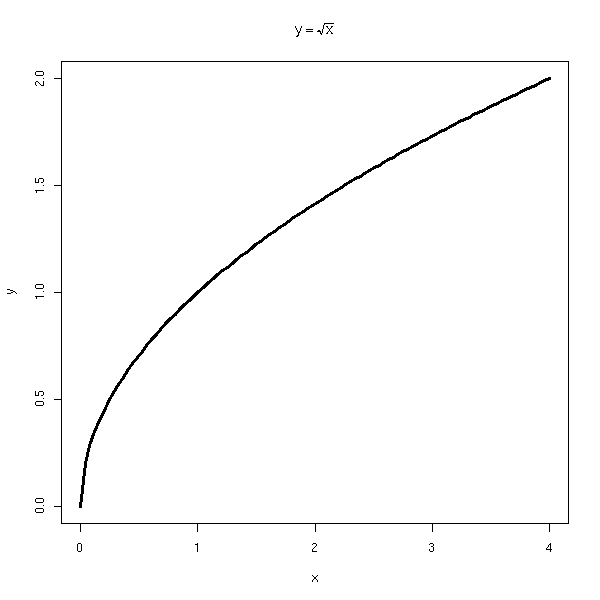
Il y a plus de détails dans le manuel :
?plotmath
À la fin de chaque session, R nous demande s'il faut sauvegarder l'environement la fois suivante : on se retrouve alors avec nos variables et fonctions (elles sont stockées dans un fichier dans le répertoire courrant ; si on fait des plusieures choses différentes avec R, il suffit d'utiliser des répertoires différents).
Ne pas oublier de nettoyer de temps à autres les variables du répertoire courrant, à l'aide des commandes ls et rm.
ls() rm(x, y, z)
On peut aussi stocker du code dans un fichier et le rappeler à l'aide de la commande source.
source("MonProgramme.R")
Il existe aussi des paquetages pour relier R à des bases de données (MySQL, PostgreSQL, Oracle, etc.) -- mais je ne les ai jamais utilisés.
Ça fonctionne d'ailleurs dans les deux sens : on peut aller chercher des données dans une base de données depuis un programme sous R, mais on peut aussi utiliser R comme langage pour les procédures stockées de PostgreSQL.
http://linuxfr.org/2003/02/20/11415.html http://archives.postgresql.org/pgsql-general/2003-02/msg00989.php http://www.joeconway.com/plr/
On les trouve sur CRAN (Complete R Archive Network)
http://cran.r-project.org/
et on les installe ainsi :
R CMD INSTALL vcd_0.1-3.tar.gz
Quand on constate qu'elles ne marchent pas, on peut les retirer ainsi :
R CMD REMOVE vcd
Quoiqu'en dise le manuel, R n'est pas un langage orienté objet.
Il y a néanmoins un peu de polymorphisme, implémenté comme suit. À chaque objet est associé une liste d'attributs, dont les classes auxquelles appartient l'objet. Ainsi, si on appelle la fonction plot avec un argument de classes "foo" et "bar", la commande va d'abord chercher une fonction plot.foo, puis une fonction plot.bar puis enfin, en cas d'échec, appellera la fonction plot.defaut. Il en va de même avec la fonction print. On peut ainsi définir ses propres « classes » et surcharger les fonctions plot et print.
> print.foo <- function (x,...) {
print.default(x)
print.default(min(x))
print.default(median(x))
print.default(max(x))
}
> x <- matrix( rnorm(20), nrow=4 )
> print(x)
[,1] [,2] [,3] [,4] [,5]
[1,] 0.05858332 -0.3082483 1.08259617 -0.10539949 -0.3734017
[2,] 0.23264808 -0.4763760 -0.01989608 -0.07837898 2.3640196
[3,] 0.05239833 -0.6764430 -0.76649216 0.76078938 0.2715206
[4,] 0.27780672 -0.5458009 -0.96929622 0.90089157 1.7325063
> attr(x, "class") <- "foo"
> print(x)
[,1] [,2] [,3] [,4] [,5]
[1,] 0.05858332 -0.3082483 1.08259617 -0.10539949 -0.3734017
[2,] 0.23264808 -0.4763760 -0.01989608 -0.07837898 2.3640196
[3,] 0.05239833 -0.6764430 -0.76649216 0.76078938 0.2715206
[4,] 0.27780672 -0.5458009 -0.96929622 0.90089157 1.7325063
attr(,"class")
[1] "foo"
[1] -0.9692962
[1] 0.01625113
[1] 2.364020On peut aussi définir ses propres fonctions surchargeables ainsi :
print <- function (x, ...) UseMethod("print")
La notion centrale est celle de méthode, et non pas celle d'objet...
Il n'y a pas du tout d'encapsulation...
Pour compliquer les choses, il existe deux manières de définir et manipuler les objets : l'ancienne (que je viens d'exposer brièvement) et la nouvelle :
library(help=methods) http://www.omegahat.org/RSMethods/Intro.ps
Je ne détaille pas, car seule la syntaxe change : la notion centrale reste celle de méthode, pas celle d'objet -- à n'utiliser que pour faire des choses vraiment génériques, par exemple surcharger les méthodes print, summary ou plot.
A FAIRE : d'après l'exemple suivant (à commenter), qui vient de bioconductor, c'est différent...
library('methods')
setClass('microarray', ## the class definition
representation( ## its slots
qua = 'matrix',
samples = 'character',
probes = 'vector'),
prototype = list( ## and default values
qua = matrix(nrow=0, ncol=0),
samples = character(0),
probes = character(0)))
dat = read.delim('../data/alizadeh/lc7b017rex.DAT')
z = cbind(dat$CH1I, dat$CH2I)
setMethod('plot', ## overload generic function `plot'
signature(x='microarray'), ## for this new class
function(x, ...)
plot(x@qua, xlab=x@samples[1], ylab=x@samples[2], pch='.', log='xy'))
ma = new('microarray', ## instantiate (construct)
qua = z,
samples = c('brain','foot'))
plot(ma)Si vous voulez regarder d'un peu plus près la programmation orientée objets sous R, le plus simple est de regarder les bibliothèques qui l'utilisent, comme pixmap (dans toutes celles qui sont installées ici, c'est la seule...).
less /usr/lib/R/library/pixmap/R/pixmap.R
Autres exemples (j'ai fini par installer tout ce qui était sur CRAN) :
MASS/scripts/ch03.R DBI gpclib pixmap SparseM
Pour des exemples plus gros :
http://www.bioconductor.org/
Pour des raisons d'efficacité, tout le code d'une bibliothèque ne sera pas écrit sous R : les parties du programme qui requièrent plus de temps (ou de mémoire), i.e., les goulots d'étranglement numérique seront écrites dans un langage plus rapide d'exécution (généralement le C, mais je crois qu'on trouve encore des gens qui programment en Fortran -- pourtant nous sommes au 21ième siècle !).
La procèdure à suivre est expliquée dans le document "Writing R extensions". Je me contente de reprendre leur exemple :
A FAIRE : vérifier que ça marche
Dans un fichier foobar.c :
void convolve(double *a, int *na, double *b, int *nb, double *ab)
{
int i, j, nab = *na + *nb - 1;
for(i = 0; i < nab; i++)
ab[i] = 0.0;
for(i = 0; i < *na; i++)
for(j = 0; j < *nb; j++)
ab[i + j] += a[i] * b[j];
}
Créer une bibliothèque partagée (si c'est pour un paquetage, c'est
automatique) :
R CMD SHLIBS foobar.c
Charger la bibliothèque partagée : pour un paquetage, on utiliserait
.First.lib <- function(lib, pkg) {
library.dynam("foobar",pkg,lib)
cat("...")
}
mais pour une utilisation ponctuelle :
dyn.load("foobar")
On peut alors l'utiliser ainsi :
conv <- function(a, b)
.C("convolve",
as.double(a),
as.integer(length(a)),
as.double(b),
as.integer(length(b)),
ab = double(length(a) + length(b) - 1))$abMentionnons aussi la fonction .Call, qui permet d'utiliser des types de données plus complexes (le source C utilise R.h et Rinternals.h ou Rdefines.h ; la fonction C prend des objets de type SEXP en argument et renvoie un objet de type SEXP), et la fonction .External (avec un seul argument, qui contient la liste des arguments).
Je ne donne pas plus de détails et me contente de renvoyer à "Writing R extensions".
(Signalons qu'on peut aussi appeler du code R depuis le C.)
Ici aussi, ça fonctionne dans les deux sens : on peut appeler du code Perl depuis R (par exemple, pour utiliser le réseau ou des expressions régulières) ou appeler R depuis Perl.
A FAIRE : donner un exemple d'appel de R depuis Perl.
R::initR("--silent", "--vanilla");
my @x = 1..100;
R::callWithNames("plot", { x => \@x, ylab => 'foo bar' });
R::eval("plot(1:10)");
Quand on manipule des données très volumineuses (domaine dans lequel R n'est justement pas très bon), on peut les stocker dans une base de données : ainsi, on se contente de récupérer et de mettre en mémoire les données sur lesquelles on calcule réellement.
A FAIRE
Dans SPlus, il y a une fonction .Java, permettant d'appeler du code Java, mais visiblement pas dans R.
A FAIRE : vérifier (c'est peut-être un paquetage que je n'ai pas installé). A FAIRE : expliquer comment contourner cette restriction. Peut-on accéder à JNDI/RMI depuis Perl ???
Vincent Zoonekynd
<zoonek@math.jussieu.fr>
latest modification on Wed Oct 13 22:32:31 BST 2004